复旦大学余宏杰课题组在基于基因数据的传染病时空溯源领域发表综述
近期,复旦大学余宏杰课题组在基于基因数据的传染病时空溯源领域发表重要综述,以“Approaches and challenges to inferring the geographical source of infectious disease outbreaks using genomic data”为题,于11月29日在线发表在The Lancet Microbe(论文链接:https://www.thelancet.com/journals/lanmic/article/PIIS2666-5247(23)00296-3/fulltext)。
近年来,新发和再发传染病的暴发流行严重威胁全球公共卫生安全。理解传染病发生、发展的暴发流行动态对疫情防控至关重要,包括疫情何时出现、何时输入、从哪里输入、是否得到有效控制等一系列科学问题。经典的流行病学调查可提供报告病例的旅行史和暴露史等信息,但很难捕获疾病在第一例病例报告前的潜隐传播动态。结合病原体基因组数据和流行病学数据可更加全面揭示传染病暴发的传播动态和时空溯源。余宏杰课题组前期定量评估了全球SARS-CoV-2基因监测的异质性和基因数据的共享程度(Chen Z et al, Nature Genetics 2022)。在此基础上,为更深入了解近年来传染病时空溯源研究方法的进展,进行相关技术和能力的储备,课题组又进一步系统总结了当前利用病原体基因组数据研究传染病时空溯源的主要研究方法和面临的挑战,强调了结合病原体基因组、流行病学监测和人群移动等多源数据,在新发和再发传染病暴发时快速、准确提供传播动态线索的重要性。
近年来,在测序技术成本不断降低的背景下,基于基因数据的系统发生学(phylogenentic)和系统发生生物地理学(phylogeography)的研究方法得到不断改进,在新冠大流行期间发挥了重要作用。虽然已有研究总结了基因流行病学和系统动力学方法在新冠大流行早期的作用(Hill V et al, Trends in parasitology 2021; Attwood SW et al, Nature reviews Genetics 2022),但未聚焦在时空溯源领域,也没有充分强调病原体基因组数据抽样和共享等关键技术环节的挑战。
通过文献系统综述,我们发现目前用于传染病暴发时空溯源的主要方法包括祖先性状重建法(ancestral trait reconstruction)和结构群落法(structured population)。祖先性状重建法包括最大简约、最大似然和贝叶斯法;结构群落法则分为结构溯祖法(structured coalescent)和结构出生-死亡法(structured birth-death)(图1)。本综述概述了各种研究方法的原理和优缺点,进一步强调了其近些年在传染病暴发溯源领域的具体应用。
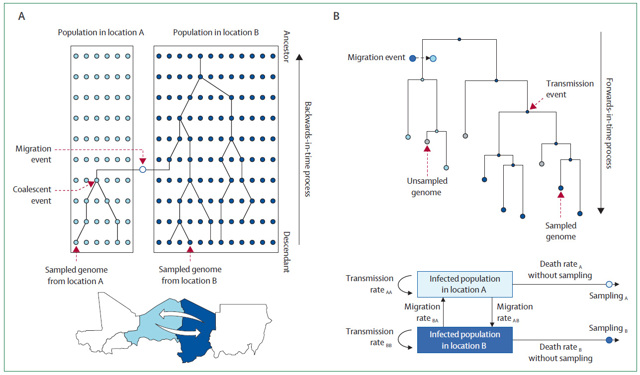
图1. 两种结构群落法的示意图
随后,综述概述了目前此领域所面临的挑战,包括:1)暴发时期对测序技术的需求;2)差异化的基因监测方案;3)基因组数据的及时和充分共享;4)暴发序列如何抽样;5)全球背景序列如何抽样;6)方法学本身的局限性(图2)。

图2. 目前此领域面临的主要挑战
本综述有助于理解在真实世界研究中如何进行方法的选择,如何解读分析结果,提高了我们对基因组数据如何在传染病时空溯源领域发挥作用的认识。随着重大、新发传染病在全球的不断暴发流行,利用日益增多的基因组数据可快速为公共卫生决策提供科学依据。此外,本综述概述了此领域所面临的主要挑战,也为未来基因监测方案的改善和方法学的研究提供了潜在方向。
复旦大学公共卫生学院2022级博士生陈志元为第一作者,复旦大学公共卫生学院教授、上海市重大传染病和生物安全研究院和复旦大学附属华山医院感染病科双聘PI余宏杰为通讯作者。本研究得到国家自然科学基金委(82130093、82073613)等项目的资助。